核心提示: 9月28日,我校金梅林教授团队联合军事科学院军事医学研究院赵志虎研究组、英国爱丁堡大学Grzegorz Kudla组和武汉菲沙基因等团队在SARS-CoV-2 RNA基因组结构动态变化上的研究成果以题为“In vivo structure and dynamics of the SARS-CoV-2 RNA genome”发表于国际期刊Nature Communications。

南湖新闻网讯(通讯员 黄坤)9月28日,我校金梅林教授团队联合军事科学院军事医学研究院赵志虎研究组、英国爱丁堡大学Grzegorz Kudla组和武汉菲沙基因等团队在SARS-CoV-2 RNA基因组结构动态变化上的研究成果以题为“In vivo structure and dynamics of the SARS-CoV-2 RNA genome”发表于国际期刊Nature Communications。
该研究全面绘制了SARS-CoV-2在整个生命周期的体内RNA-RNA相互作用,提供了转录调节序列(TRS-L 和 TRS-Bs)之间相互作用的第一个直接证据,发现其基因组 RNA 经历了强烈的压缩,但基因组结构域保持稳定,结构域特征信号增强,全局环化减弱。该研究为病毒复制、不连续转录和翻译移码的调节提供了结构基础,描述了SARS-CoV-2生命周期中RNA结构的动态变化,将有助于人们开发出更好的抗病毒策略。
研究者鉴定出了对于SARS-Cov-2复制和不连续转录至关重要的5'-UTR的全部五个保守茎环结构(SL1-SL5),并且在3' stem-loop II-like motif (S2M)区域鉴别到一个替代的3'-UTR结构。在互作矩阵中,研究者发现了SARS-CoV-2基因组5'-end和3'-end之间的相互作用,确定了基因组环化(genome cyclization)现象并推测其与复制/包装有关。
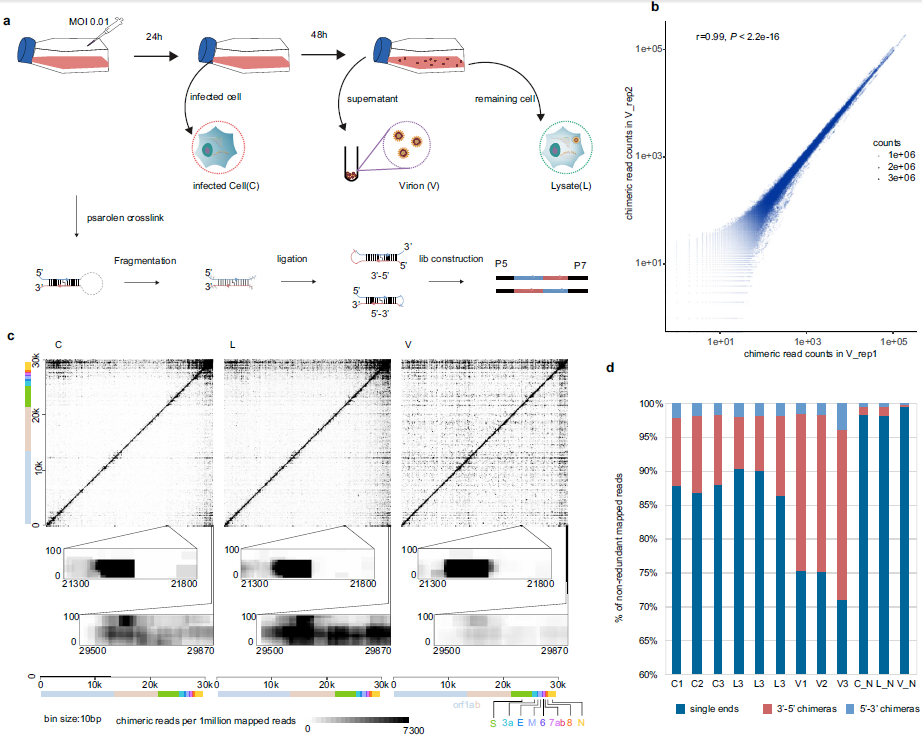
图1 实验设计和不同感染阶段SARS-CoV-2的RNA互作图谱
RNA邻近连接反应能得到RNA-RNA相互作用信息,其中包含一部分剪切本之间的连接,也包含sgRNA自身及其与宿主RNA因空间邻近而连接的嵌合分子。为了正确识别RNA-RNA相互作用,研究者评估了已发布的SARS-CoV-2感染细胞RNA-Seq数据集,确定了三个特征来将RNA-Seq中发现的大多数sgRNA嵌合体与真正的邻近连接嵌合体区分开来:sgRNAs几乎完全以5'-3'方向连接,而RNA邻近连接嵌合体可以为5'-3'方向和3'-5'方向;嵌合体臂之间的连接点精确定位在sgRNAs中,而邻近连接中的连接位点是可变的;sgRNA嵌合体通常包括嵌合体的TRS-L和TRS-B侧之间的同源区域 ,而邻近连接嵌合体通常不包括这样的区域。
研究者通过调整分析流程参数可以检测或去除RNA-Seq数据中大多数sgRNA嵌合体,使用严谨参数流程来分析邻近连接数据,同时过滤掉sgRNA。通过分析5'-3'和 3 '-5'嵌合体以识别由TRS-L介导的RNA-RNA相互作用,并分析TRS-L连接位点的分布,发现可以区分实验数据中TRS-L介导的RNA-RNA相互作用和TRS-L依赖的sgRNA。远程相互作用介导的RNA碱基配对表明TRS-L可能与TRS-B区域稳定结合。有趣的是, TRS-L通常不与确切的TRS-B序列相互作用,而是与50nt内的侧翼序列相互作用,这可能是为下一步配对Ccs-B和模板转换提供灵活性。
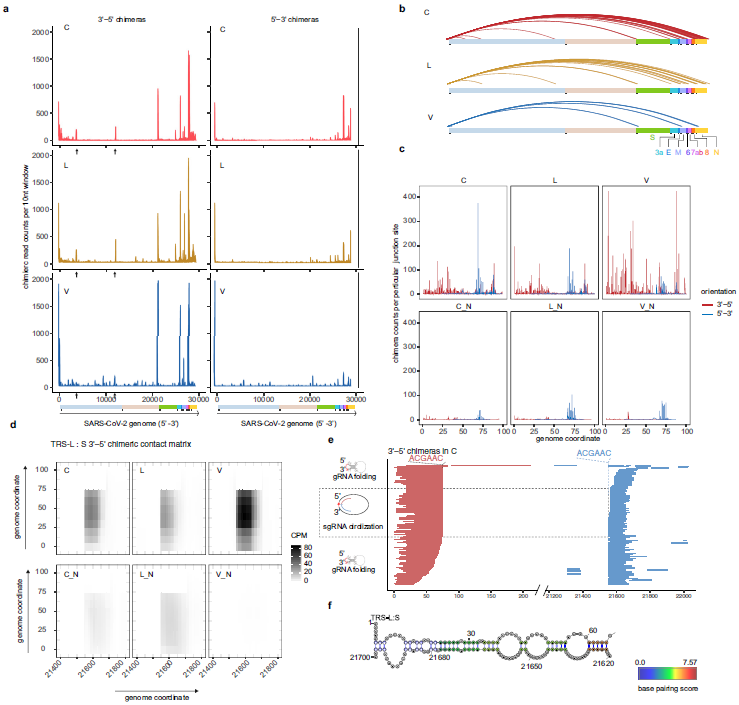
图2 TRS-L与经典TRS-B位点的互作
除了经典的sgRNA,研究者还观察到与TRS-L相互作用的其他区域,并通过RT-PCR和Sanger测序验证TRS-L介导的相互作用会产生新的候选sgRNA。这些新的sgRNA没有经典的ACGAAC 核心序列motif,但与其存在部分overlap,表明TRS-L和负链上的部分cCS-B的碱基配对对于这些sgRNA不连续转录过程中的模板转换至关重要。
SARS-Cov-2的一个特征是使用程序化的核糖体移码来促进ORF1b编码RdRp的翻译并控制其蛋白质的相对表达。研究者分析了移码元件(FSE)的相互作用,不仅发现了之前已经报道的三茎假结结构,还发现了更大的嵌有FSE的稳定茎环(arch1)的可选择局部结构,以及多种由FSE介导的远程相互作用,并鉴定得到多个强远程互作,利用三维建模对其进行了可视化。
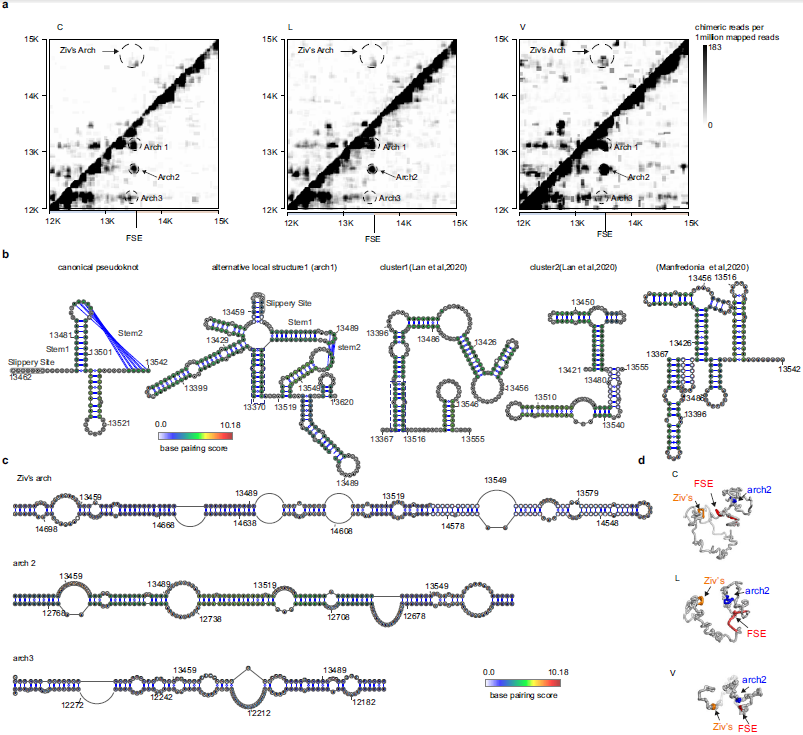
图3 局部可变及远程的FSE结构
研究者通过相关性分析、主成分分析和差异分析展示了病毒生命周期各个阶段的互作动态,结果表明,与细胞组和裂解组相比,病毒衣壳中SARS-CoV-2基因组的三分之一的相互作用密度较低、基因组环化减少,然而病毒粒子和裂解细胞中近端和长程相互作用在增加,意味着基因组在包装成病毒粒子期间发生压缩,TRS-L区域介导的相互作用的变化则可能反映了病毒基因组包装的第一步。研究者还发现,病毒粒子长距离loop的差异互作中,减弱的相互作用几乎总是由sgRNA介导,而L组较C组也出现这种现象,表明RNA相互作用和构象在病毒包装过程中逐渐发生变化,sgRNAs丰度可能在减少。
简化的SPLASH数据热图类似于哺乳动物基因组Hi-C数据 ,研究者应用insulation score算法检查SARS-CoV-2基因组RNA是否被划分为domain,以及基因组RNA的整体压缩是否会导致domain的破坏,结果发现在基因组压缩和包装期间,domain结构不仅保留了,而且还得到了加强。最后,研究者通过计算SARS-CoV-2基因组的Shannon entropy value发现,细胞组比病毒粒子中的RNA有更灵活的结构,结构域边界的灵活性,可能是更有吸引力的药物设计靶点。
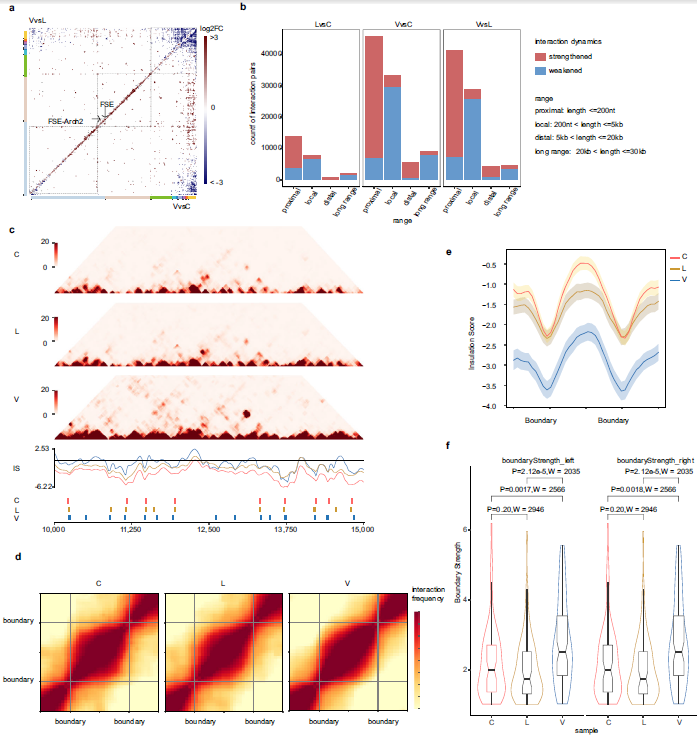
图4 SARS-CoV-2生命周期不同阶段的RNA互作动态
该研究简化了SPLASH方法,捕获了多个阶段的SARS-CoV-2中RNA-RNA相互作用,观察到了RNA经典和替代结构,包括 5'-UTR 和 3'-UTR、移码元件 (FSE) 假结和基因组环化,揭示了FSE附近的短距离和长距离arch形成嵌入FSE的“高阶假结”, 描述了SARS-CoV-2生命周期中RNA结构的动态变化,为其复制、不连续转录和翻译移码的调节提供了结构基础,将有助于人们开发出更好的抗病毒策略。
我校动科动医学院博士研究生黄坤、军事医学研究院张彦以及武汉菲沙基因谢德健为该论文的共同第一作者,我校金梅林教授、军事医学研究院赵志虎研究员、英国爱丁堡大学Grzegorz Kudla研究员为共同通讯作者。
原文链接:https://www.nature.com/articles/s41467-021-25999-1
审核人 金梅林
