核心提示: 近期,曹罡教授与戴金霞副研究员团队开发了新型空间组学技术MiP-seq,该项研究成果在线发表于学术期刊Nature Biomedical Engineering杂志。
南湖新闻网讯(通讯员 吴小凤)近期,曹罡教授与戴金霞副研究员团队开发了新型空间组学技术MiP-seq,该技术是基于成像的空间多组学双端原位测序技术(图1),打破了传统技术瓶颈,以亚细胞、单碱基分辨率同时实现了DNA、RNA、蛋白质和功能小分子的多维空间组学3D图谱,可以兼容转录组、蛋白组、拉曼成像、钙离子成像多个模态信息检测。该项研究成果以“Spatial multi-omics at subcellular resolution via high-throughput in situ pairwise sequencing”为题,于5月14日在线发表于学术期刊Nature Biomedical Engineering杂志。
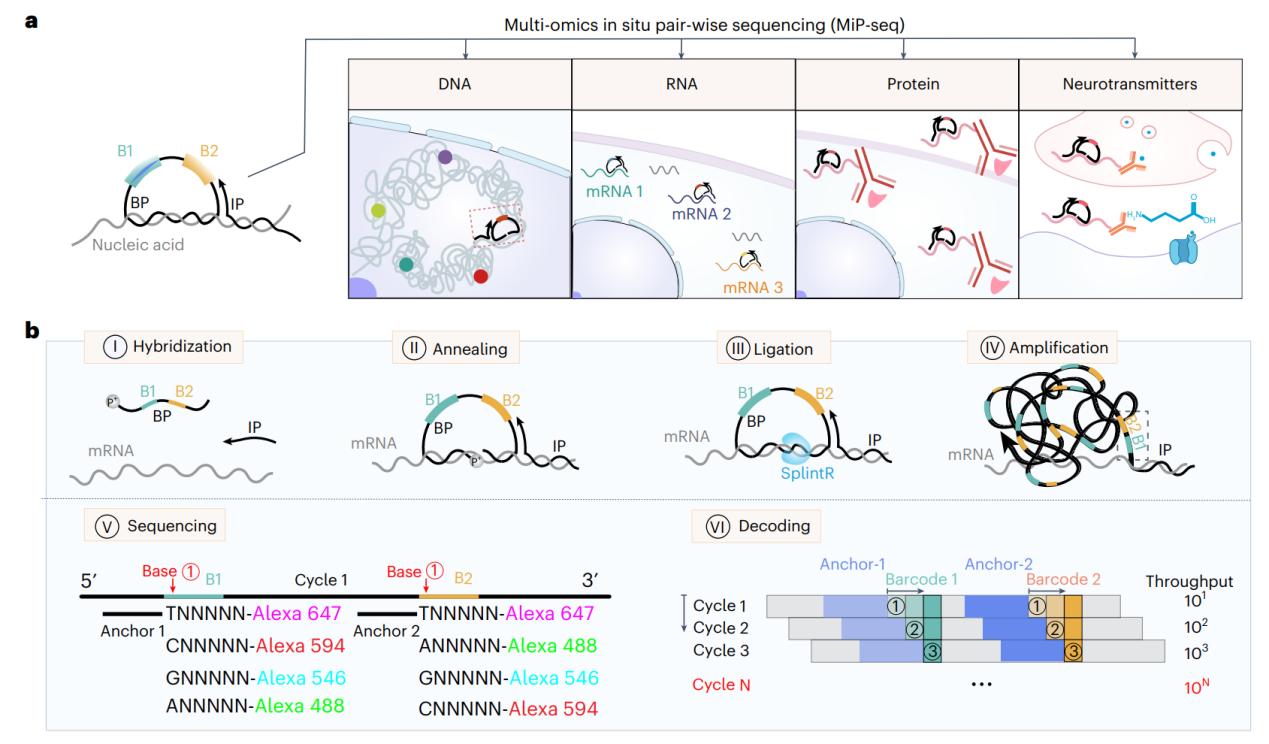
图1:MiP-seq的操作原理及流程示意图
解析组织空间异质性及其分子空间表达模式的空间组学是生命科学研究的关键前沿技术,其多组学拓展广度与高通量检测深度已为各研究领域的热点问题带来了新的发现与见解。空间组学方法主要分为基于二代测序(Next generation sequencing, NGS)捕获或成像策略两类。基于NGS的空间组学方法可进行全基因组、转录组范围的捕获,且部分方法可拓展至多组学,但缺乏单细胞分辨率,而且检测效率较低、成本较高、批间差异较大,重构的空间组学图像实质为2D马赛克间断式平面图片;基于成像的空间组学方法具有亚细胞分辨率,且捕获效率高,部分方法兼容多组学,可利用成像实现3D无间断空间组学,但大多数方法为靶向空间组,全转录组模式捕获效率较低,多轮成像光毒性大,高密度信号解析困难。因此各种空间组学方法都亟待进一步升级,以满足科研对空间组真正单细胞高分辨、高捕获率、高重复性的迫切需求。
MiP-seq的主要创新点有五点,一是MiP-seq采用双条形码锁式探针设计和双端测序策略。与传统的单条形码锁式探针和单端测序策略相比,本方法具有更高的解码能力(10N vs 4N),并且需要的测序轮数显著减少。因此MiP-seq可以大幅降低测序和成像成本,并且可以节省约50%的成本及测序时间。同时与单条码和单端测序相比,双条码测序策略可以显著缩短成像时间,从而最大限度地减少光毒性;二是MiP-seq成功实现了原位多组学检测(图2),以亚细胞分辨率同时描绘了同一切片中DNA、RNA和蛋白质的空间景观以及神经递质的空间分布。这种综合性的空间多组学方法以往未被报道过;三是光信号拥挤是几乎所有基于成像空间组学方法的“瓶颈”问题。MiP-seq提出了一种序列稀释策略,通过使用一组测序引物和锚定基因引物来解决信号拥挤问题,将极大地促进高密度信号的精确解析;四是与当前的原位测序方法相比,MiP-seq的探针设计策略及生化反应体系显著提高了原位测序检测效率。同时,MiP-seq可以达到单核苷酸分辨率来检测 DNA 和 RNA 的单碱基突变、单核苷酸多态,并可以区分 RNA m6A 修饰。基于 MiP-seq的高通量和单碱基精度,小鼠海马中亲代基因的等位基因空间特异性表达模式得以解析;五是MiP-Seq与拉曼成像和活体Ca2+成像相结合,获得了具有多维信息的组织图谱,对解析细胞表达谱与功能的关联十分必要(图3)。
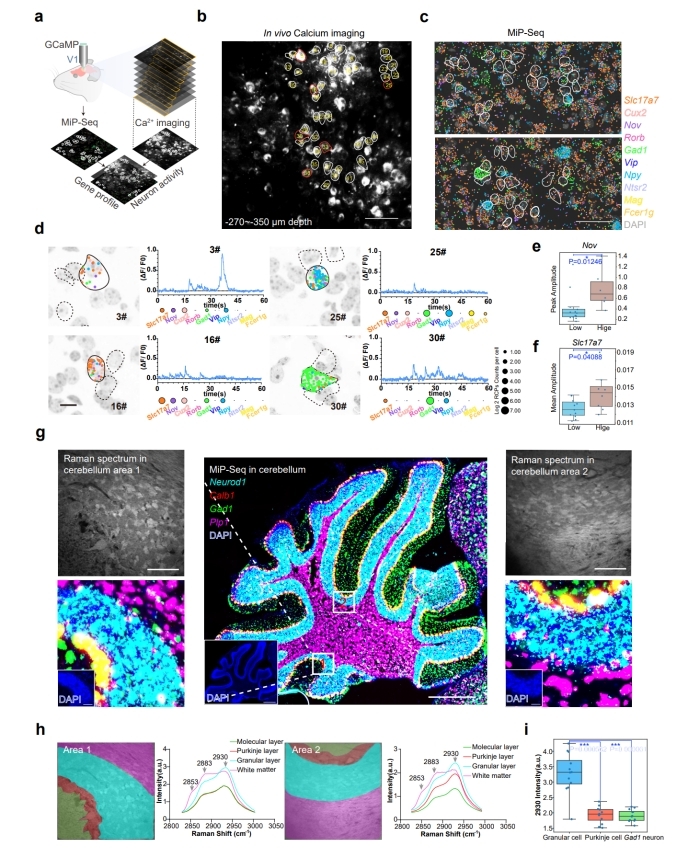
图2:应用MiP-seq解析弥漫性大B淋巴瘤组织样本中30种蛋白及6种mRNA的空间分布
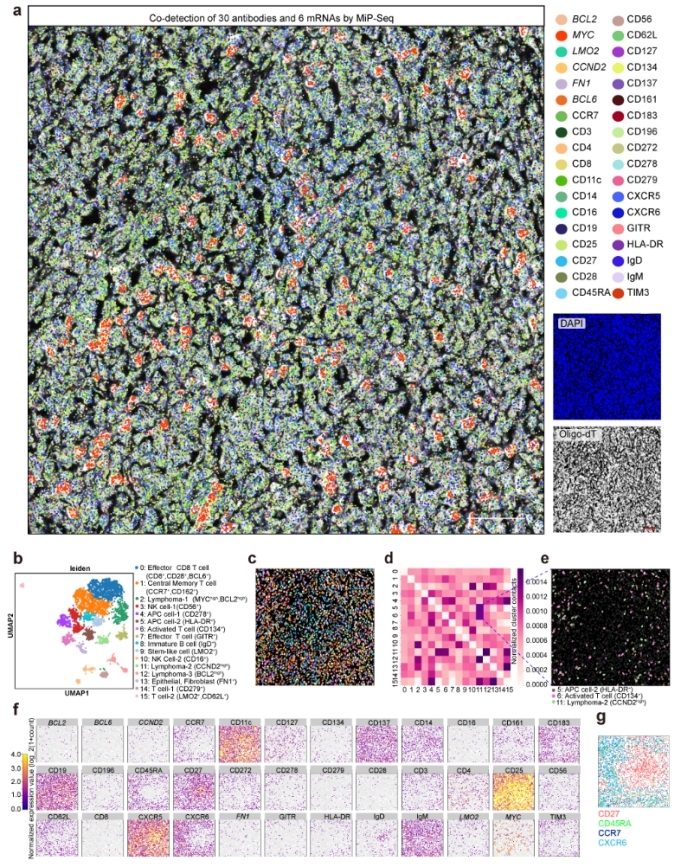
图3:MiP-seq与体内Ca2+成像和拉曼成像整合分析
综上所述,MiP-seq方法在捕获策略、解码通量、靶标检测范围、高密度成像、SNV、RNA修饰、功能成像整合分析等方面进行了优化与提升。相较于现行的空间组学方法有其独特的优势,在靶向空间多组学细分领域具有可观的应用价值及前景。这种高检测效率的空间多组学方法将为发育生物学、脑科学、免疫学、肿瘤学、微生物学等领域提供更精准的高空间分辨率的多维空间图谱。以生物分子的实景还原方式来解析细胞行为与功能的方式更精准地揭示了生命进程。本技术已实现产业转化(鲲羽生物https://www.spatialfish.com/),将为基础科学研究与临床分子病理诊断提供高效的空间多组学服务。
据悉,曹罡教授、戴金霞副研究员为该论文共同通讯作者,我校吴小凤博士后和徐伟泽博士生为该论文的共同第一作者。该工作得到了国家自然科学基金委创新群体项目、联合基金项目和面上项目的资助。
审核人 戴金霞
【英文摘要】
Technology for spatial multi-omics aids the discovery of new insights into cellular functions and disease mechanisms. Here we report the development and applicability of multi-omics in situ pairwise sequencing (MiP-seq), a method for the simultaneous detection of DNAs, RNAs, proteins and biomolecules at subcellular resolution. Compared with other in situ sequencing methods, MiP-seq enhances decoding capacity and reduces sequencing and imaging costs while maintaining the efficacy of detection of gene mutations, allele-specific expression and RNA modifications. MiP-seq can be integrated with in vivo calcium imaging and Raman imaging, which enabled us to generate a spatial multi-omics atlas of mouse brain tissues and to correlate gene expression with neuronal activity and cellular biochemical fingerprints. We also report a sequential dilution strategy for resolving optically crowded signals during in situ sequencing. High-throughput in situ pairwise sequencing may facilitate the multidimensional analysis of molecular and functional maps of tissues.
