核心提示: 北京时间2023年6月14日23点,Nature(《自然》)杂志发表了华中农业大学李国田教授团队牵头完成的题为“Genome editing of a rice CDP-DAG synthase confers multipathogen resistance”的研究成果。该研究成果对扩大抗病基因来源,推动作物抗病育种、植物病害绿色防控,保障国家粮食安全具有重要意义。
南湖新闻网讯(通讯员 沙干)北京时间2023年6月14日23点,Nature(《自然》)杂志发表了华中农业大学李国田教授团队牵头完成的题为“Genome editing of a rice CDP-DAG synthase confers multipathogen resistance”的研究成果。该团队克隆到一个广谱抗病类病斑突变体基因RBL1,并通过基因编辑创制了增强作物广谱抗病性且稳产的新基因RBL1Δ12,该基因在作物中高度保守,与传统抗病基因相比,可打破物种界限、普适性更强,具有巨大抗病育种应用潜力。

水稻是全球重要的粮食作物之一,生产上常受稻瘟病、稻曲病、白叶枯病等多种病害威胁。稻瘟病被称为“水稻癌症”,广泛分布于世界各稻区,是一种毁灭性的真菌病害,全球每年因稻瘟病造成的产量损失达数千万吨。培育种植广谱抗病品种是实现稻瘟病绿色防控最经济有效的措施,对保障我国和全球的粮食安全具有重要意义。类病斑突变体材料可在没有病原菌侵染的情况下自发产生超敏反应坏死斑,具有广谱抗病性,但产量较低,限制了类病斑突变体基因在作物抗病育种中的应用。
华中农大团队前期联合加州大学戴维斯分校等研究团队,在全基因组测序的水稻突变体库中筛选到一个对稻瘟病菌和白叶枯菌都具有良好抗性的类病斑突变体株系rbl1,其突变基因RBL1编码一个胞苷二磷酸-二酰甘油合成酶。体外补充磷脂酰肌醇及过表达磷脂酰肌醇合成酶基因OsPIS1可部分回补rbl1突变体表型,表明RBL1基因通过调控磷脂酰肌醇的生物合成调控细胞程序性死亡和免疫。
进一步分析显示,rbl1突变体细胞膜PI(4,5)P2含量较野生型显著减少。同时发现,水稻PI(4,5)P2在稻瘟病菌侵染时被招募到侵染菌丝周围,并在稻瘟病菌效应蛋白分泌结构中富集,证明PI(4,5)P2在水稻-稻瘟病菌互作中发挥重要作用(图一)。可进一步推测,抑制或破坏病原菌特异侵染结构的形成可能是平衡产量和免疫的一种新策略。
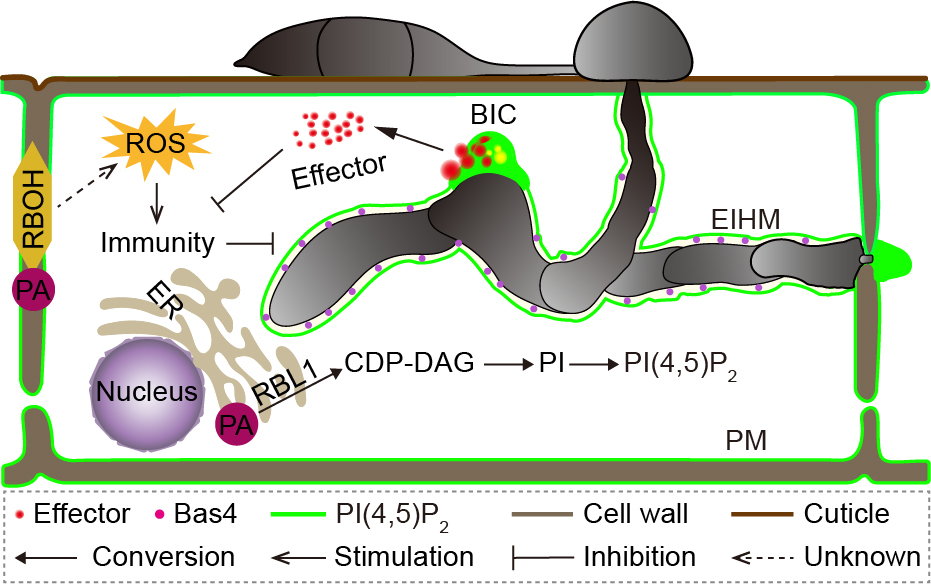
图一 RBL1工作模型
rbl1类病斑突变体株系虽具广谱抗病性但产量极低,研究团队通过对RBL1基因编码区多位点进行编辑,创制了一个新基因RBL1Δ12,rbl1Δ12株系只在成株期呈现微弱的类病斑表型。研究发现,RBL1Δ12基因显著增强了水稻对不同地区分离的10个稻瘟菌、5个白叶枯菌和2个稻曲菌生理小种的抗性。大田试验分析发现,rbl1Δ12株系稳产且具有显著的抗稻瘟病能力,在稻瘟病害严重发生时能够挽救约40%产量损失(图二)。
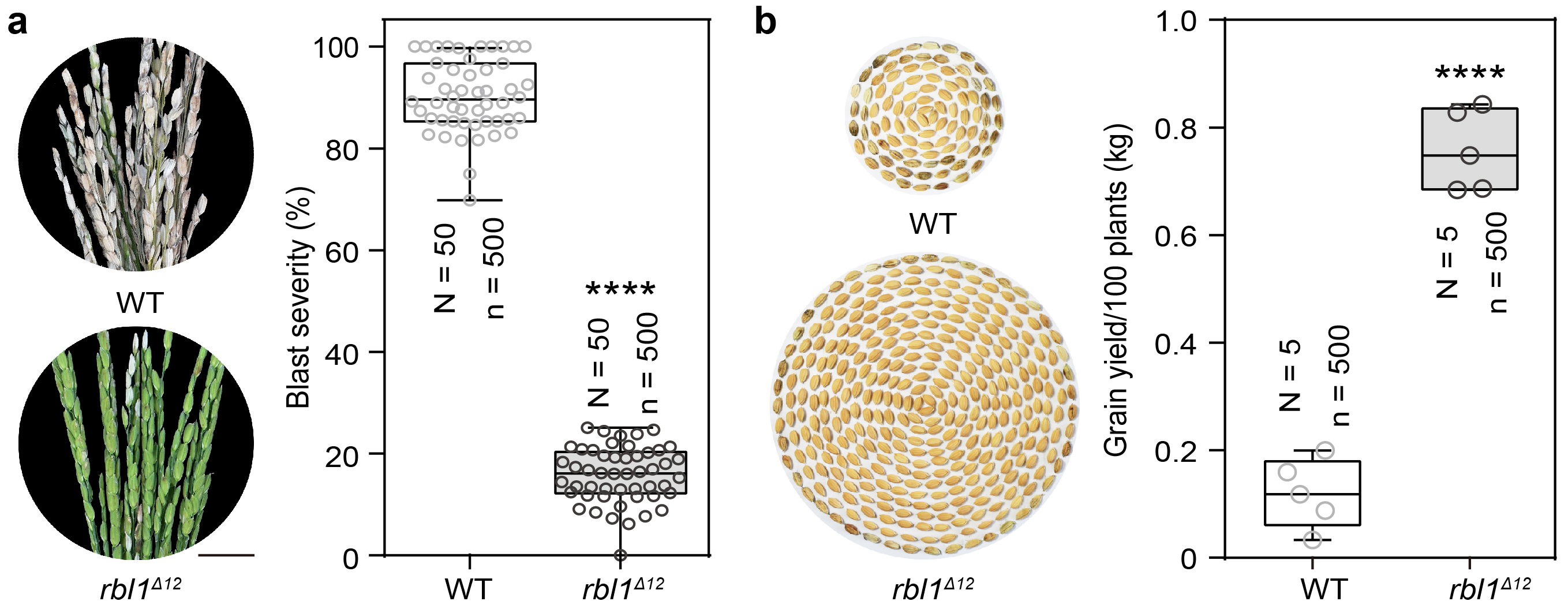
图二 rbl1Δ12株系抗病性强且产量稳定
该基因在作物中高度保守,经研究团队初步测试,该基因在小麦抗锈病和纹枯病上也有显著效果,进一步证明其在作物抗病育种中的巨大应用潜力,该研究模式也为类病斑突变体储备资源的利用提供了借鉴。该研究成果对扩大抗病基因来源,推动作物抗病育种、植物病害绿色防控,保障国家粮食安全具有重要意义。
值得一提的是,Nature杂志特邀作者以Research Briefing形式对该成果进行科普性报道,并得到国内外同行高度评价。
农业微生物资源发掘与利用全国重点实验室、湖北洪山实验室李国田教授和加州大学戴维斯分校、劳伦斯伯克利国家实验室Pamela C. Ronald院士为该论文共同通讯作者。李国田教授团队博士研究生沙干、孙鹏为本论文共同第一作者,课题组多位学生共同参与。该研究得到国家重点研发、自然科学基金、中央高校基本科研业务费专项资金、杂交水稻国家重点实验室开放研究基金等的资助。西北农林科技大学康振生院士、法国波尔多大学Yohann Boutté教授、作物遗传改良全国重点实验室郭亮教授、李强教授和谢卡斌教授、江西省农业科学院水稻研究所黄仁良副研究员、农业微生物资源发掘与利用全国重点实验室郑露副教授、深圳华大生命科学研究院丘璨瑜副研究员、山东省农科院徐建第研究员、澳大利亚阿德莱德大学Jenny Mortimer教授等国内外科研机构研究人员参与了合作研究。
【英文摘要】
The discovery and application of genome editing introduced a new era of plant breeding by giving researchers efficient tools for the precise engineering of crop genomes1. Here we demonstrate the power of genome editing for engineering broad-spectrum disease resistance in rice (Oryza sativa). We first isolated a lesion mimic mutant (LMM) from a mutagenized rice population. We then demonstrated that a 29-base-pair deletion in a gene we named RESISTANCE TO BLAST1 (RBL1) caused broad-spectrum disease resistance and showed that this mutation caused an approximately 20-fold reduction in yield. RBL1 encodes a cytidine diphosphate diacylglycerol synthase that is required for phospholipid biosynthesis2. Mutation of RBL1 results in reduced levels of phosphatidylinositol and its derivative phosphatidylinositol 4,5-bisphosphate (PtdIns(4,5)P2). In rice, PtdIns(4,5)P2 is enriched in cellular structures that are specifically associated with effector secretion and fungal infection, suggesting that it has a role as a disease-susceptibility factor3. By using targeted genome editing, we obtained an allele of RBL1, named RBL1Δ12, which confers broad-spectrum disease resistance but does not decrease yield in a model rice variety, as assessed in small-scale field trials. Our study has demonstrated the benefits of editing an LMM gene, a strategy relevant to diverse LMM genes and crops.
论文链接: https://www.nature.com/articles/s41586-023-06205-2
审核人 李国田
